医学成像中的深度学习——基于PyTorch的3D 医学图像分割
计算机视觉领域深度网络的兴起为经典图像处理技术表现不佳的问题提供了最先进的解决方案。在图像识别的广义任务中,包括目标检测、图像分类和分割、活动识别、光流和姿态估计等问题,我们可以很容易地声称 DNN(深度神经网络)取得了卓越的性能。
深度学习和医学成像
计算机视觉领域深度网络的兴起为经典图像处理技术表现不佳的问题提供了最先进的解决方案。在图像识别的广义任务中,包括目标检测、图像分类和分割、活动识别、光流和姿态估计等问题,我们可以很容易地声称 DNN(深度神经网络)取得了卓越的性能。
随着计算机视觉的兴起,人们对医学成像领域的应用产生了浓厚的兴趣。尽管医学成像数据不是那么容易获得,但 DNN 似乎是对如此复杂和高维数据进行建模的理想候选者。
正如我们将看到的,医学图像通常是 3 维或 4 维的。该领域引起广泛关注的另一个原因是它对人类生活的直接影响。在美国,医疗差错是继心脏病和癌症之后的第三大死亡原因。因此,很明显,人类死亡的前三个原因与医学成像有关。这就是为什么预计到2023 年,医学影像中的人工智能和深度学习将创造一个超过 10 亿美元的全新市场。
3D医学图像分割的需求
医学图像中的3D 立体图像分割对于诊断、监测和治疗计划是必不可少的。我们将只使用磁共振图像 (MRI)。手动练习需要解剖知识,而且费用昂贵且耗时。另外,由于人为因素,它们可能不准确。尽管如此,自动体积分割可以节省医生的时间,并为进一步分析提供准确的可重复解决方案。
我们将从描述 MR 成像的基础开始,因为了解输入数据对于训练深度架构至关重要。然后,我们为读者提供可有效用于此任务的 3D-UNET 概述。
医学影像和核磁共振
医学成像旨在揭示隐藏在皮肤和骨骼中的内部结构,以及诊断和治疗疾病。医学磁共振(MR) 成像使用来自氢原子核的信号来生成图像。在氢核的情况下:当它暴露于外部磁场(表示为 B0)时,磁矩或自旋会像罗盘指针一样与磁场方向对齐。
所有恒定磁化强度都通过一个额外的射频脉冲旋转到另一个平面,该脉冲足够强并且施加的时间足够长以倾斜磁化强度。励磁后,磁化立即在另一个平面上旋转。旋转磁化在接收线圈中产生 MR 信号。然而,MR 信号由于两个独立的过程而迅速衰减,这两个过程会降低磁化强度,从而导致返回到激发前存在的稳定状态,从而产生所谓的T1 图像和 T2 磁共振图像. T1 弛豫与原子核对其周围的能量过剩有关,而 T2 弛豫是指各个磁化矢量开始相互抵消的现象。上述现象是完全独立的。因此,不同的强度代表不同的组织,如下图所示:
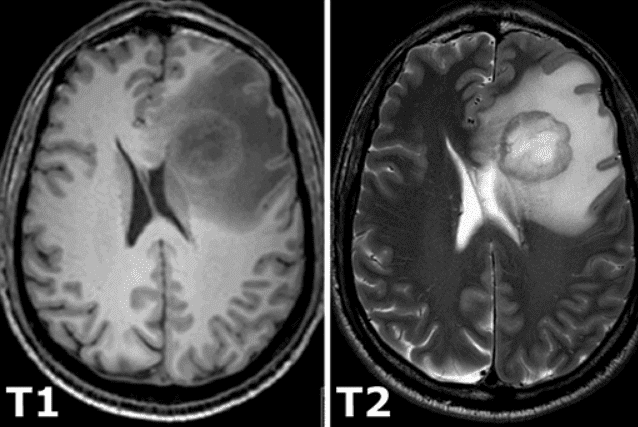
3D 医学图像表示
由于医学图像表示 3D 结构,因此处理它们的一种方法是使用 3D 体积的切片并执行常规的 2D 滑动卷积,如下图所示。假设红色矩形是一个 5x5 的图像块,可以用包含强度值的矩阵表示。体 素强度和核用 3x3 卷积核进行卷积,如下图所示。在相同的模式下,内核在整个 2D 网格(医学图像切片)上滑动,并且每次我们执行互相关。卷积的 5x5 补丁的结果存储在 3x3 矩阵中(出于说明目的没有填充),并在网络的下一层传播。
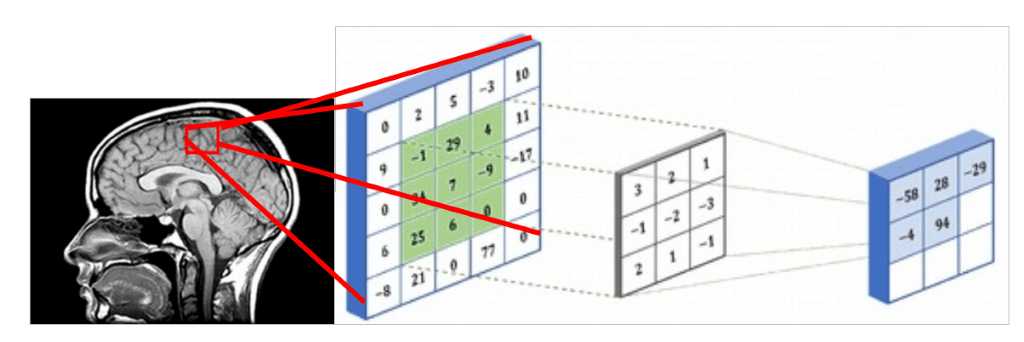
或者,您可以将它们表示为类似于中间层的输出。在深度架构中,我们通常有多个特征图,这实际上是一个 3D 张量。如果有理由相信附加维度之间存在模式,则执行 3D 滑动卷积是最佳选择。医学图像就是这种情况。与编码 2D 域中对象的空间关系的 2D 卷积类似,3D 卷积可以描述 3D 空间中对象的空间关系。由于2D 表示对于医学图像来说是次优的,我们将在这篇文章中选择不使用 3D 卷积网络。

医学图像切片可以看作是一个中间层的多个特征图,不同的是它们之间有很强的空间关系
模型:3D-Unet
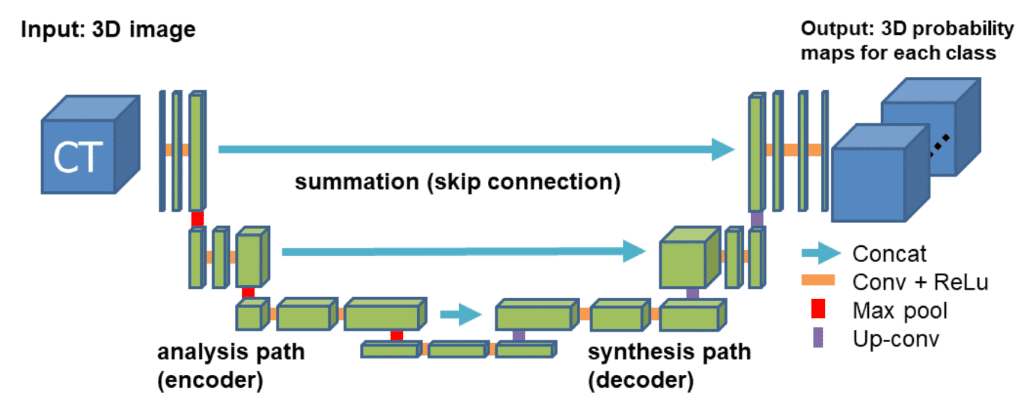
对于我们的示例,我们将使用广为接受的 3D U 形网络。后者(代码)扩展了对称 u 形 2D Unet 网络的连续概念,该网络在 RGB 相关任务(例如语义分割)中产生了令人印象深刻的结果。该模型有一个编码器(收缩路径)和一个解码器(合成路径)路径,每个路径都有四个分辨率步骤。在编码器路径中,每一层包含两个 3 ×3 ×3 卷积,每个卷积后面跟着一个整流线性单元 (ReLu),然后是一个 2 ×2 ×2 最大池化,每个维度的步幅为 2。在解码器路径中,每一层由一个 2×2×2 的转置卷积组成,每个维度的步幅为 2,然后是两个 3×3×3 的卷积,每个卷积后面是一个 ReLu。快捷方式跳过连接来自分析路径中相同分辨率的层为合成路径提供了必要的高分辨率特征。在最后一层,一个 1×1×1 的卷积将输出通道的数量减少到标签的数量。通过在最大池化之前将通道数加倍来避免瓶颈。在每个 ReLU 之前引入了3D批量归一化。每个批次在训练期间使用其平均值和标准差进行归一化,并使用这些值更新全局统计数据。接下来是一个明确学习规模和偏差的层。下图说明了网络架构。
损失函数:Dice loss
由于固有的任务不平衡,交叉熵不能总是为这个任务提供好的解决方案。具体来说,交叉熵损失会单独检查每个像素,将类别预测(深度像素向量)与我们的 one-hot 编码目标向量进行比较。因为交叉熵损失单独评估每个像素向量的类别预测,然后对所有像素进行平均,所以我们本质上是在对图像中的每个像素进行平等学习。如果您的各种类在图像中的表示不平衡,这可能是一个问题,因为最流行的类可以主导训练。
我们将尝试在脑部 MRI 中区分的 4 类在图像中具有不同的频率(即空气比其他组织具有更多的实例)。这就是采用骰子损失度量的原因。它基于Dice 系数,它本质上是两个样本之间重叠的度量。该度量的范围从 0 到 1,其中 Dice 系数为 1 表示完全重叠。Dice loss 最初是为二进制分类而开发的,但它可以推广到与多个类一起工作。随意使用我们的 Dice loss 的多类实现。
医学影像数据
深度架构需要大量训练样本才能产生任何有用的广义表示,而标记的训练数据通常既昂贵又难以产生。这就是为什么我们每天都会看到使用生成学习来生成越来越多的医学成像数据的新技术。此外,训练数据必须能够代表网络将来会遇到的数据。如果训练样本是从与现实世界中遇到的不同的数据分布中提取的,那么网络的泛化性能将低于预期。
由于我们专注于大脑 MRI 自动分割,因此简要描述 DNN 试图区分的大脑基本结构很重要 a) 白质 (WM),b)灰质 (GM),c) 脑脊液 (CSF ) . 下图说明了脑 MRI 切片中的分割组织。

2017 年 I-Seg 医学影像数据挑战赛
在这个关键时期将婴儿大脑MRI 图像准确分割为白质 (WM)、灰质 (GM) 和脑脊液 (CSF),对于研究正常和异常的早期大脑发育都具有重要意义。生命的第一年是出生后人类大脑发育最活跃的阶段,伴随着快速的组织生长和广泛的认知和运动功能的发展。这个早期阶段对许多神经发育和神经精神疾病至关重要,例如精神分裂症和自闭症。这一关键时期越来越受到人们的关注。
该数据集旨在促进 6 个月婴儿脑部 MRI 的自动分割算法。本次挑战赛与MICCAI 2017联合举办,共有21支国际团队参赛。该数据集包含 10 个来自专家的密集注释图像和 13 个用于测试的图像。不提供测试标签,您只有在官网上传结果后才能看到您的分数。对于每个主题,都有一个 T1 加权和 T2 加权图像。
第一个主题将用于测试。原始 MR 卷的大小为 256x192x144。在 3D-Unet 中,使用的采样子体积大小为 128x128x64。生成的训练数据集由 500 个子卷组成。对于验证集,使用了来自一名受试者的 10 个随机样本。
Medical Zoo Pytorch
为什么:我们的目标是在 PyTorch 中实现最先进的 3D 深度神经网络的 开源医学图像分割库以及最常见的医学数据集的数据加载器。我们存储库的第一个稳定版本预计将很快发布。
我们坚信开放和可重复的深度学习研究。为了重现我们的结果,此存储库中提供了这项工作的代码和材料。该项目最初是一篇硕士论文,目前正在进一步开发中。
把它们放在一起-实施细节
我们使用了 PyTorch 框架,它被认为是最广泛接受的深度学习研究工具。随机梯度下降,单个批次大小,学习率为 1e-3,权重衰减为 1e-8,用于所有实验。我们在存储库中提供了测试,您可以轻松地重现我们的结果,以便您可以使用代码、模型和数据加载器。
最近我们用 Pytorch 添加了 Tensorboard 可视化。这个惊人的功能可以让您保持清醒,并让您跟踪模型的训练过程。下面你可以看到一个保持训练统计数据的例子,骰子系数。和损失以及每类分数以了解模型行为。
代码
让我们将所有描述的模块放在一起,用 MedicalZoo 用一个简短的脚本(用于说明目的)设置一个实验。
# Python libraries
import argparse
import os
# Lib files
import lib.medloaders as medical_loaders
import lib.medzoo as medzoo
import lib.train as train
import lib.utils as utils
from lib.losses3D import DiceLoss
def main():
args = get_arguments()
utils.make_dirs(args.save)
training_generator, val_generator, full_volume, affine = medical_loaders.generate_datasets(args,
path='.././datasets')
model, optimizer = medzoo.create_model(args)
criterion = DiceLoss(classes=args.classes)
if args.cuda:
model = model.cuda()
print("Model transferred in GPU.....")
trainer = train.Trainer(args, model, criterion, optimizer, train_data_loader=training_generator,
valid_data_loader=val_generator, lr_scheduler=None)
print("START TRAINING...")
trainer.training()
def get_arguments():
parser = argparse.ArgumentParser()
parser.add_argument('--batchSz', type=int, default=4)
parser.add_argument('--dataset_name', type=str, default="iseg2017")
parser.add_argument('--dim', nargs="+", type=int, default=(64, 64, 64))
parser.add_argument('--nEpochs', type=int, default=200)
parser.add_argument('--classes', type=int, default=4)
parser.add_argument('--samples_train', type=int, default=1024)
parser.add_argument('--samples_val', type=int, default=128)
parser.add_argument('--inChannels', type=int, default=2)
parser.add_argument('--inModalities', type=int, default=2)
parser.add_argument('--threshold', default=0.1, type=float)
parser.add_argument('--terminal_show_freq', default=50)
parser.add_argument('--augmentation', action='store_true', default=False)
parser.add_argument('--normalization', default='full_volume_mean', type=str,
help='Tensor normalization: options ,max_min,',
choices=('max_min', 'full_volume_mean', 'brats', 'max', 'mean'))
parser.add_argument('--split', default=0.8, type=float, help='Select percentage of training data(default: 0.8)')
parser.add_argument('--lr', default=1e-2, type=float,
help='learning rate (default: 1e-3)')
parser.add_argument('--cuda', action='store_true', default=True)
parser.add_argument('--loadData', default=True)
parser.add_argument('--resume', default='', type=str, metavar='PATH',
help='path to latest checkpoint (default: none)')
parser.add_argument('--model', type=str, default='VNET',
choices=('VNET', 'VNET2', 'UNET3D', 'DENSENET1', 'DENSENET2', 'DENSENET3', 'HYPERDENSENET'))
parser.add_argument('--opt', type=str, default='sgd',
choices=('sgd', 'adam', 'rmsprop'))
parser.add_argument('--log_dir', type=str,
default='../runs/')
args = parser.parse_args()
args.save = '../saved_models/' + args.model + '_checkpoints/' + args.model + '_{}_{}_'.format(
utils.datestr(), args.dataset_name)
return args
if __name__ == '__main__':
main()实验结果
下面你可以看到模型的训练和验证骰子损失曲线。监控您的模型性能并调整参数以获得如此平滑的训练曲线非常重要。很容易理解这个模型的效率。
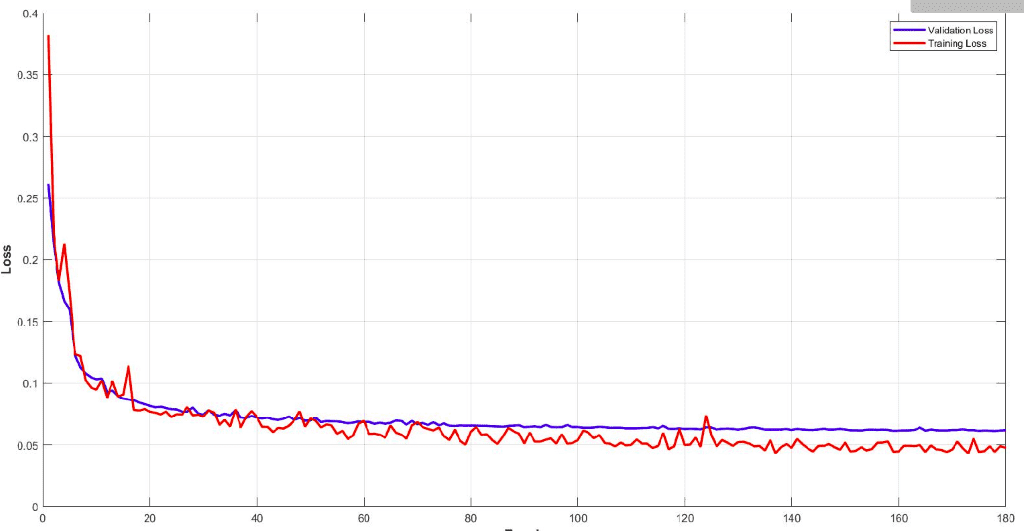
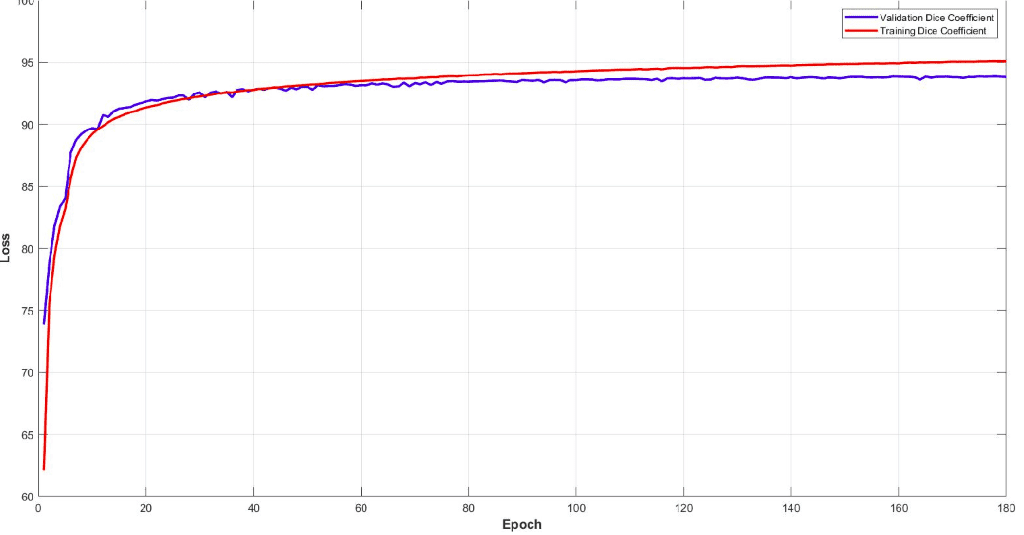
该模型在子卷的验证集中达到了大约 93% 的骰子系数分数。最后但同样重要的是,让我们看看验证集中 3D-Unet 的一些可视化预测。尽管预测是 3D 体积,但我们在这里只展示一个代表性切片。通过获取 MRI 的多个子体积,可以将它们组合起来形成完整的 3D MRI 分割。请注意,我们使用子卷采样的事实是数据增强。

来自训练有素的 3D-Unet 的非标准化最后一层预激活。网络学习高度语义的任务相关内容,这些内容对应于类似于输入的大脑结构。

我们的预测 VS 基本事实。您认为哪个预测是基本事实?仔细看看再做决定!需要注意的是,我们在这里只展示了中轴切片,但预测是一个 3D 体积。可以观察到,该网络完美地预测了空气体素,但难以区分组织边界。但是,让我们再次检查以找出真正的那个!
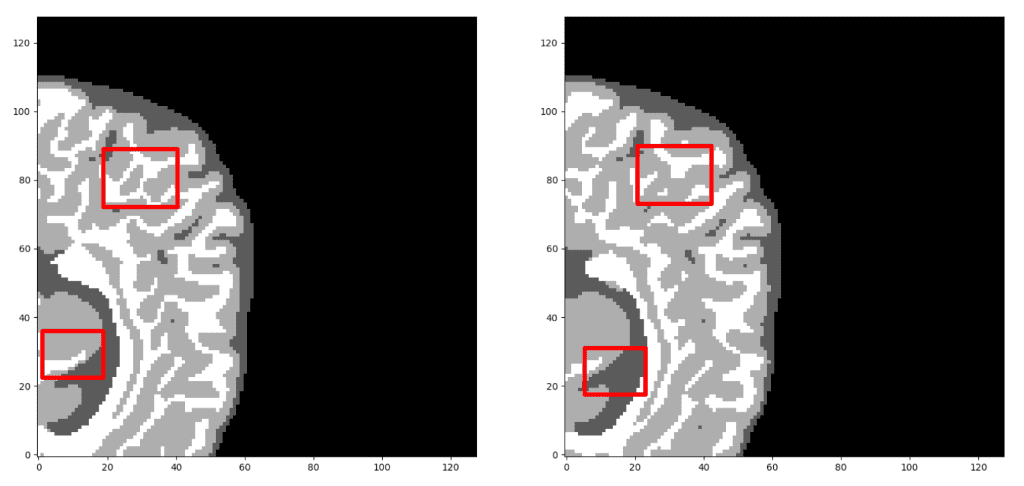
现在,我相信您可以区分基本事实。如果不确定,请查看文章末尾:)
最近我们还使用 Pytorch 添加了Tensorboard可视化。这个惊人的功能可以让你保持清醒,让你跟踪模型的训练过程。您可以在下面看到一个保留训练统计数据、骰子系数和损失以及每类分数以了解模型行为的示例。

很明显,不同的组织具有不同的精度,即使从训练开始也是如此。例如,查看验证集中从高值开始的空气体素,因为它是不平衡数据集的最主要类别。另一方面,灰质从最低值开始,因为它最难区分且训练实例较少。
结论
这篇文章部分说明了我们团队开发的MedicalZoo Pytorch 库的一些特性。深度学习模型将为社会提供身临其境的医学影像解决方案。
在本文中,我们回顾了医学成像和 MRI 的基本概念,以及如何在深度学习架构中表示和使用它们。然后,我们描述了一种被广泛接受的高效 3D 架构(Unet)和骰子损失函数来处理类不平衡。最后,我们结合了上述所有特征,并使用库脚本提供了我们在脑 MRI 中的实验分析的初步结果。结果证明了 3D 架构的效率和深度学习在医学图像分析中的潜力。
翻译原稿来自:https://theaisummer.com/medical-image-deep-learning/
iSeg-2017 challenge:https://iseg2017.web.unc.edu/
MedicalZooPytorch:https://github.com/black0017/MedicalZooPytorch
更多推荐
 已为社区贡献1条内容
已为社区贡献1条内容









所有评论(0)