如何在GEO数据库上传原始测序数据RNA-SEQ,学习过程分享!
为了让大家更好的利用公共数据库中的数据,所以需要大家对自己上传的数据进行充分的注释,geo官方给定的了一个metadata spreadsheet (template and examples),(https://submit.ncbi.nlm.nih.gov/geo/submission/meta/)里面包含了RNA-seq和ChIP-seq数据的模板,可以根据模板填入。待所有数据上传完成并提交
背景知识
GEO (Gene Expression Omnibus)是一个公共功能基因组学数据存储库,支持符合 MIAME 标准的数据提交。 接受基于阵列和序列的数据。 提供工具来帮助用户查询和下载实验和策划的基因表达谱。基本上文章中原始测序数据都会提交在这个数据库中,所以学会上传自己的数据也是十分重要的。
2 上传步骤
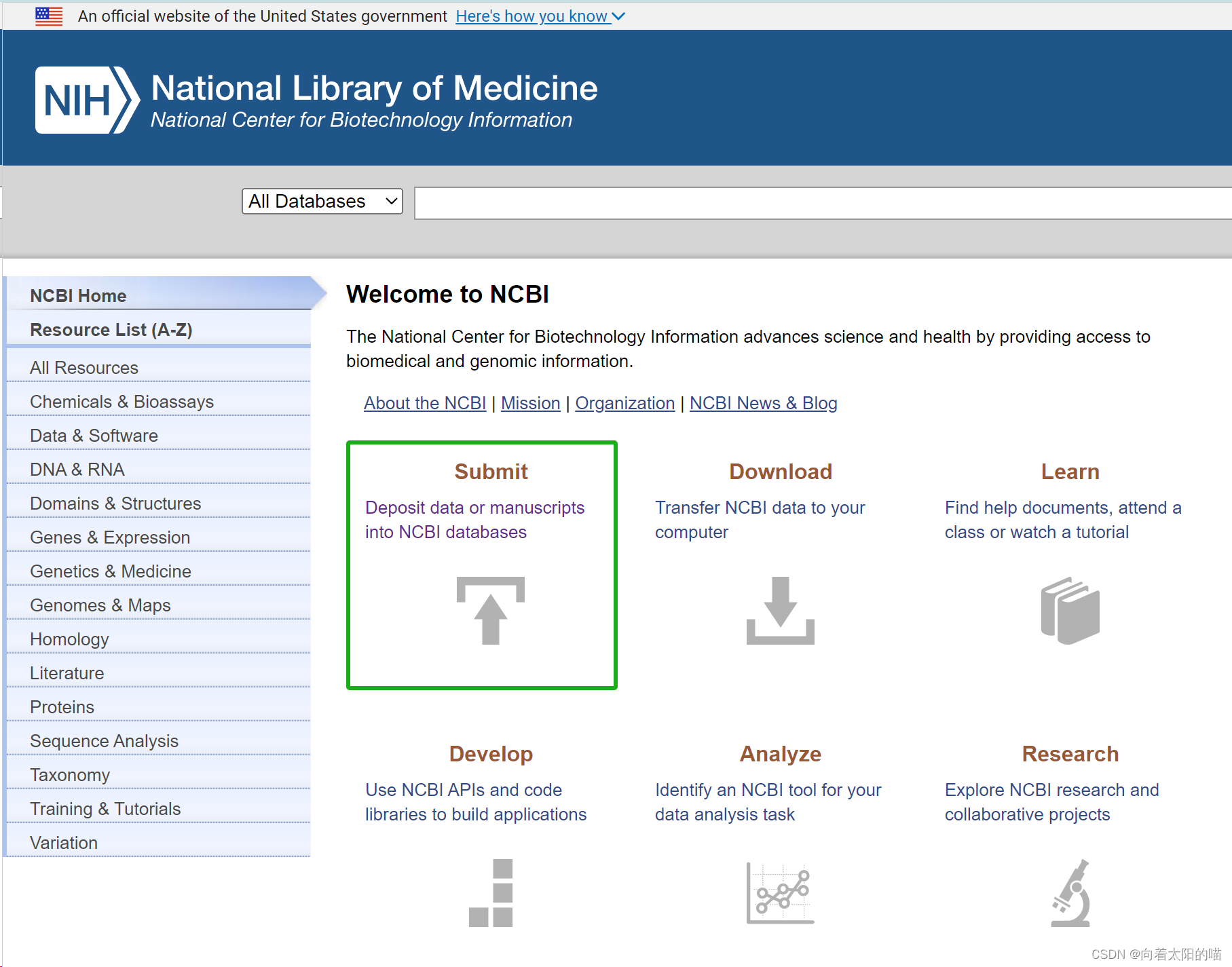
1.登录NCBI(https://www.ncbi.nlm.nih.gov/),在首页点击Submit,准备上传原始测序数据(.fastq.gz文件,是测序原始文件的压缩文件)。
2.下滑页面,找到Other Tools下面的GEO一栏,点击Learn more 3.点击Sunbmit high-throughput sequencing
3.点击Sunbmit high-throughput sequencing
4.点击Raw data files
5.上传数据分为两步,页面往下滑动,先选择Transfer Files 6.登录NCBI账号(需要自己注册)
6.登录NCBI账号(需要自己注册)
上传数据是会要求填入自己的邮件,工作单位和地址等信息。
7.上传数据,Step1提供了文件上传地址,Step2提供了FTP服务器的地址,账号和密码。
8.选择远程登录FTP服务器软件filezilla并上传测序文件
在给定的上传文件夹中新建一个文件夹(命名规则:geo_submission_month&day),并把测序原始文件从上传到此文件夹中。eg:geo_submission_feb22
注意:右侧的uploads/…需要按回车的,我还查了半天,以为我的filezilla有问题。。。。。哈哈哈,愚蠢的错误
【正在进行中,这一步十分漫长。2个小时两个文件的龟速。。。】
今晚放这上传,如果明天还很慢,再想办法服务器传输,今天试了conda下载安装lftp,好像不太行,可能我不太会弄,哈哈哈。
9.因为远程上传文件,需要检查文件是否上传完整,所以需要同时上传md5值
#在自己的服务器中将所有的原始测序文件生成原始md5值
md5sum *fastq.gz >md5check.txt
10.测序数据注释和实验流程概述
为了让大家更好的利用公共数据库中的数据,所以需要大家对自己上传的数据进行充分的注释,geo官方给定的了一个metadata spreadsheet (template and examples),(https://submit.ncbi.nlm.nih.gov/geo/submission/meta/)里面包含了RNA-seq和ChIP-seq数据的模板,可以根据模板填入。填完之后就随数据同时上传即可。
下图是metadata的示例,根据自己的数据情况,按照模版填写即可。
11.等所有数据上传完成后,就可以进入最后一步,Notify GEO。
然后填入你上传的文件夹名。如果文章在投或者暂不想公开数据,GEO数据可以将你的测序文件设为私密(最多3年),这个可以自行设置Release date。最后点击Submit,即完成上传的所有流程。
12.最后一步~邮件确认!
待所有数据上传完成并提交之后,会有邮件提醒你的上传是否存在问题,如果无误,就会给定GSE number,表示上传完成。
撒花!!!出GEO编号啦,大概需要一天的样子,我周五晚上还在上传,周六就给GEO编号啦,周一早上打开电脑,一看出来啦,NCBI真给力!!!接下来就是改改论文,准备投稿啦,嘿嘿嘿。
############小结,就是需要上传的文件有3个。
1.fastq原始数据;2.表达量矩阵;3.metadata样本说明表格。
更多推荐
 已为社区贡献1条内容
已为社区贡献1条内容









所有评论(0)